반응형
새로운 문제가 오늘 따근따근하게 올라왔으니 한번 어떤가 풀어봐야겠다. 문제 풀이라기 보단 그냥 힌트로 블럭 조립하는 느낌이다.
새로운 문제의 1단계는
'Candidatus Carsonella ruddii PV'라는 세균의 전체 유전체 서열을 FASTA 포맷으로 가져오고 코딩서열 (CDS) 목록을 포함하는 유전체의 주석 정보를 GenBank flat file 포맷으로 가져오기 바랍니다.
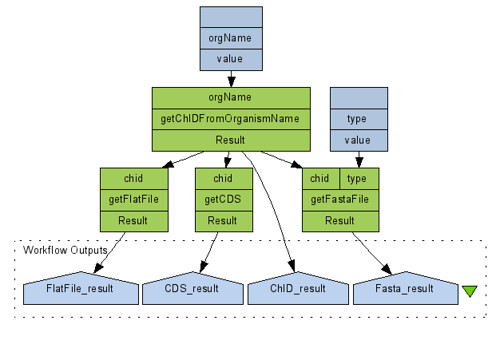
DDBJ에서 제공하는 GIB 데이터베이스의 웹 서비스를 이용하면 된다. 우선 Candidatus Carsonella ruddii PV 라는 세균의 GIB에서 제공하는 ChID를 알아야 이 세균에 대한 정보를 얻을 수 있다. 따라서 이름을 가지고 ChID를 얻어오는 getChIDFromOrganismName을 사용한다. getChIDFromOrganismName는 GIBV(http://xml.nig.ac.jp/wsdl/GIBV.wsdl)와 Gib( http://xml.nig.ac.jp/wsdl/Gib.wsdl)에 둘다 존재한다. GIBV는 Viruses에 대한 데이터베이스이고, Gib는 microbial genomes에 대한 데이터베이스이다. 문제로 출제된 'Candidatus Carsonella ruddii PV'라는 세균을 계속해서 GIBV의 getChIDFromOrganismName으로 찾으니 나오나!!!
다행히 Gib의 getChIDFromOrganismName으로 ChID(chromosome ID)가 Crud_PV: 임을 알아내고 바로 이 결과를 getFastaFile(type인자로 genome을 주고)로 보내서 전체 서열에 대한 FASTA포맷의 결과를 얻어냈다.
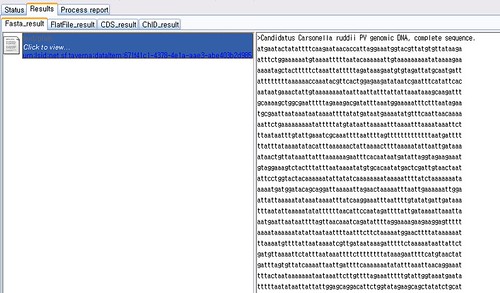
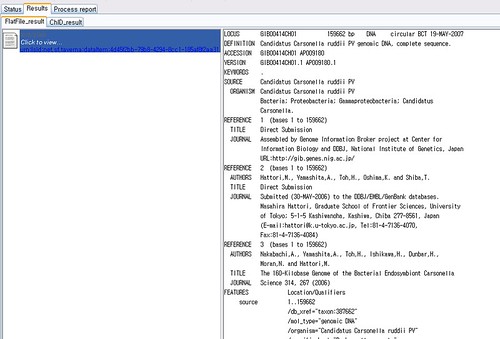
이제 코딩서열(CDS) 목록을 포함하는 유전체의 주석정보를 가지고 오면 된다. 그것도 GenBank Flat File형태로 이건 getFlatFile을 사용했다. getFlatFile과 getFastaFile은 모두 ChID를 인자로 주어야 한다. 그외 CDS 정보만 얻길 원한다면 getCDS를 통해서 CDS정보만을 가져 올 수 있다.
두번째로는 ORF의 아미노산 서열을 찾아 multi-fasta 포맷으로 저장하는 것이다. getFasta로 얻은 전체서열에 대한 ORF는 EBI에서 제공하는 nucleic_gene_finding의 getorf를 사용하면 된다. getorf의 sequence_direct_data를 getFasta의 fasta서열로 지정하고, sformat을 fasta로 지정하면 해당 서열에 대한 ORF를 반환한다. 결과로 얻은 multi-fasta 형식의 ORF 아미노산 서열은 18,156 라인의 8,610개의 서열에 해당하는 좀 많은?? 서열이기 때문에 이와 상동성이 높은 단백질 데이터베이스인 SwissProt에 대해 Blast를 수행하기에는 좀 많다. 그래서 문제에서도 서열이 긴 순서대로 20개정도만 선택해서 가져오기를 바라고 있다. ^^;;
여기서 또하나의 문제는 20개건 만개의 서열이건 이를 Taverna에서 Blast 수행하면 하나의 서열만 수행한다는 점이다. DDBJ에서 제공하는 simplesearch나 EBI에서 제공하는 Blast, WuBlast도 multi-fasta포맷으로 여러개의 서열을 지정해도 하나의 서열만 blast를 수행하기 때문에 Blast를 수행할때 서열을 하나씩 분리해서 List형태로 만든 다음 Blast를 수행해야 한다. 따라서 다음과 같이 BeanShell 스크립트를 만들어 multi-fasta포맷의 서열을 여러개의 List로 바꾸어 blast를 수행한다.
import java.util.*;
StringTokenizer tokenizer = new StringTokenizer(input,">");
int tokensize = tokenizer.countTokens();
String[][] output;
output = new String[tokensize][1];
int i =0;
while(tokenizer.hasMoreElements()){
output[i][0] = ">" + tokenizer.nextToken();
i = i+1;
}
위의 BeanShell 스크립트에다가 서열길이가 긴 20개만 sort해서 추출하는 코드를 추가하면 된다. 다 보여주면 재미 없으니깐 sort하는 부분은 ㅋㅋㅋ
Taverna의 List 형태로 분리된 fasta 포맷의 ORF
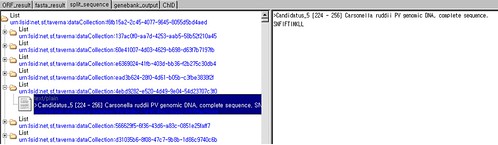
이렇게 List 향태로 분리한 후 Blast를 수행해야 모든 서열에 대해 Blast가 가능하다.
이렇게 List로 분리된 서열을 EBI의 WuBlast를 수행한다.
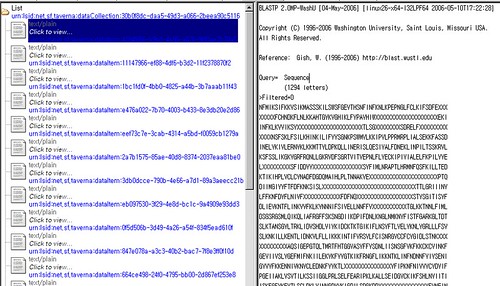
서열길이가 긴 순서대로 20개에 대해 WU-Blast를 수행한 결과는 위와 같다.
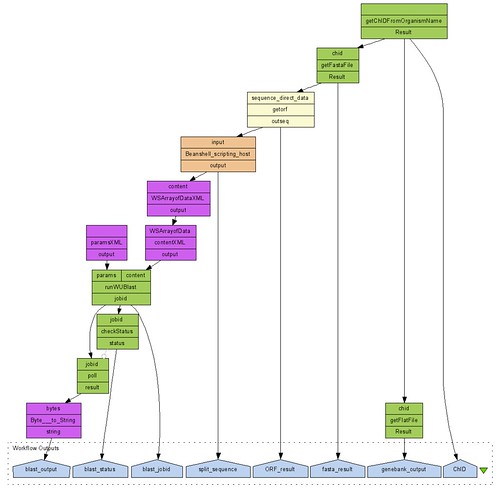
위 다이어그램은 최종적으로 작성된 다이어그램이다. 마지막으로 ORF의 정확한 시작코돈의 위치와 처음 얻은 CDS정보와 Blast의 결과를 비교하면 끝.
새로운 문제의 1단계는
'Candidatus Carsonella ruddii PV'라는 세균의 전체 유전체 서열을 FASTA 포맷으로 가져오고 코딩서열 (CDS) 목록을 포함하는 유전체의 주석 정보를 GenBank flat file 포맷으로 가져오기 바랍니다.
DDBJ에서 제공하는 GIB 데이터베이스의 웹 서비스를 이용하면 된다. 우선 Candidatus Carsonella ruddii PV 라는 세균의 GIB에서 제공하는 ChID를 알아야 이 세균에 대한 정보를 얻을 수 있다. 따라서 이름을 가지고 ChID를 얻어오는 getChIDFromOrganismName을 사용한다. getChIDFromOrganismName는 GIBV(http://xml.nig.ac.jp/wsdl/GIBV.wsdl)와 Gib( http://xml.nig.ac.jp/wsdl/Gib.wsdl)에 둘다 존재한다. GIBV는 Viruses에 대한 데이터베이스이고, Gib는 microbial genomes에 대한 데이터베이스이다. 문제로 출제된 'Candidatus Carsonella ruddii PV'라는 세균을 계속해서 GIBV의 getChIDFromOrganismName으로 찾으니 나오나!!!
다행히 Gib의 getChIDFromOrganismName으로 ChID(chromosome ID)가 Crud_PV: 임을 알아내고 바로 이 결과를 getFastaFile(type인자로 genome을 주고)로 보내서 전체 서열에 대한 FASTA포맷의 결과를 얻어냈다.

이제 코딩서열(CDS) 목록을 포함하는 유전체의 주석정보를 가지고 오면 된다. 그것도 GenBank Flat File형태로 이건 getFlatFile을 사용했다. getFlatFile과 getFastaFile은 모두 ChID를 인자로 주어야 한다. 그외 CDS 정보만 얻길 원한다면 getCDS를 통해서 CDS정보만을 가져 올 수 있다.


두번째로는 ORF의 아미노산 서열을 찾아 multi-fasta 포맷으로 저장하는 것이다. getFasta로 얻은 전체서열에 대한 ORF는 EBI에서 제공하는 nucleic_gene_finding의 getorf를 사용하면 된다. getorf의 sequence_direct_data를 getFasta의 fasta서열로 지정하고, sformat을 fasta로 지정하면 해당 서열에 대한 ORF를 반환한다. 결과로 얻은 multi-fasta 형식의 ORF 아미노산 서열은 18,156 라인의 8,610개의 서열에 해당하는 좀 많은?? 서열이기 때문에 이와 상동성이 높은 단백질 데이터베이스인 SwissProt에 대해 Blast를 수행하기에는 좀 많다. 그래서 문제에서도 서열이 긴 순서대로 20개정도만 선택해서 가져오기를 바라고 있다. ^^;;
여기서 또하나의 문제는 20개건 만개의 서열이건 이를 Taverna에서 Blast 수행하면 하나의 서열만 수행한다는 점이다. DDBJ에서 제공하는 simplesearch나 EBI에서 제공하는 Blast, WuBlast도 multi-fasta포맷으로 여러개의 서열을 지정해도 하나의 서열만 blast를 수행하기 때문에 Blast를 수행할때 서열을 하나씩 분리해서 List형태로 만든 다음 Blast를 수행해야 한다. 따라서 다음과 같이 BeanShell 스크립트를 만들어 multi-fasta포맷의 서열을 여러개의 List로 바꾸어 blast를 수행한다.
import java.util.*;
StringTokenizer tokenizer = new StringTokenizer(input,">");
int tokensize = tokenizer.countTokens();
String[][] output;
output = new String[tokensize][1];
int i =0;
while(tokenizer.hasMoreElements()){
output[i][0] = ">" + tokenizer.nextToken();
i = i+1;
}
위의 BeanShell 스크립트에다가 서열길이가 긴 20개만 sort해서 추출하는 코드를 추가하면 된다. 다 보여주면 재미 없으니깐 sort하는 부분은 ㅋㅋㅋ
Taverna의 List 형태로 분리된 fasta 포맷의 ORF

이렇게 List 향태로 분리한 후 Blast를 수행해야 모든 서열에 대해 Blast가 가능하다.
이렇게 List로 분리된 서열을 EBI의 WuBlast를 수행한다.

서열길이가 긴 순서대로 20개에 대해 WU-Blast를 수행한 결과는 위와 같다.

위 다이어그램은 최종적으로 작성된 다이어그램이다. 마지막으로 ORF의 정확한 시작코돈의 위치와 처음 얻은 CDS정보와 Blast의 결과를 비교하면 끝.
반응형