반응형
Genome Browser의 주요 기능은 1~10,000bp 또는 12,010,000~12,020,000 등등
chromosome 상에서 일정한 영역의 정보만을 DB에서 읽어와서 이를 다양한 형태로 출력해주는 것이다. 그럼 이러한 일정
영역의 데이터(feature)를 가져오는 SQL문을 만들어 보면,,,
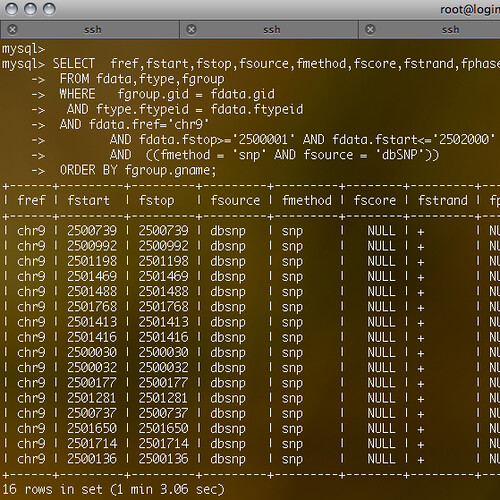
그러나 위의 SQL문은 짧은 feature가 많이 존재하거나, chromosome의 중간 부분을 검색하는 경우에 있어서는 비효율적이다. 직접 HapMap 데이터를 기준으로 chromosome 9(총 140,273,252 bp)의 2,500,001에서 2,502,000 영역에 존재하는 feature를 검색하기 위해 SQL문을 실행하면 아래와 같이 1분 3초라는 시간이 걸린다 그것도 64bit에 메모리만 12GB의 좀 좋은 사양의 서버에서,,,
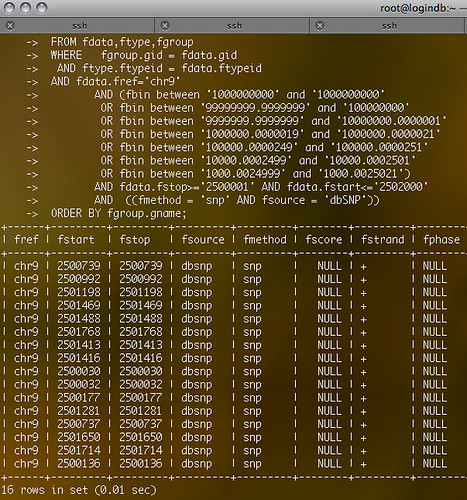
그럼 chromosome의 일정 영역에 대한 정보를 빠르게 가져올 방법은 없는 걸까? 여기에 R-Tree 알고리즘을 적용하면 400배 이상 빨라진다. 자 보시라. 0.01초
이것은 원래 지도상의 위치나 공간 정보를 다루기 위해서 만들어진 R-Tree(Guttman, a. 1984. R-Trees: A dynamic index structure for spatial searching)를 생물학 데이터에 적용한 결과이다. 공간 데이터를 가로만 있다고 하고 genome 정보를 검색하는데 사용하는 것이다.
간단하게 살펴보면, feature가 들어가는 일정한 폭의 bin(100Mb, 10Mb, 1Mb, 100Kb, 10Kb, 1Kb)에다가 float의 정수,소수 부분에 각각 폭(해당 feature가 속한 영역)과 번호(chromosome 상의 위치)를 넣게 된다. 그럼 1Kb 미만(bin=1000.012010)이고 영역이 1000(bin=10000.001201)인 feature를 검색한다면 검색의 범위가 좁아지기 때문에 훨씬 빠르게 원하는 위치의 원하는 만큼의 크기(길이)만을 가져올 수 있다(R-Tree 적용 후 그림 참고). R-tree는 gbrowse뿐만 아니라 UCSC Genome Browser에서도 사용하고 있다고 한다.
무턱대고 genome browser를 끄적 거리면서 고민했던 문제인데, 한번에 해결이 되어버리게 되었다. 좀 일찍 좀 찾아 볼 것을... 일전에 서열 유사성 검색에서 suffux tree와 함께 또하나의 Tree가 ^^;;
오늘은 요정도의 소개만 하고, 내일??은 실제 코드를 통해 어떻게 R-tree를 gbrowse에 적용해서 사용하는지에 대해서도 : )
SELECT 온갖정보들 WHERE ref="Chr1" AND start <= 12010000 AND end >= 12020000
그러나 위의 SQL문은 짧은 feature가 많이 존재하거나, chromosome의 중간 부분을 검색하는 경우에 있어서는 비효율적이다. 직접 HapMap 데이터를 기준으로 chromosome 9(총 140,273,252 bp)의 2,500,001에서 2,502,000 영역에 존재하는 feature를 검색하기 위해 SQL문을 실행하면 아래와 같이 1분 3초라는 시간이 걸린다 그것도 64bit에 메모리만 12GB의 좀 좋은 사양의 서버에서,,,
R-Tree 적용 전
그럼 chromosome의 일정 영역에 대한 정보를 빠르게 가져올 방법은 없는 걸까? 여기에 R-Tree 알고리즘을 적용하면 400배 이상 빨라진다. 자 보시라. 0.01초
R-Tree 적용 후
이것은 원래 지도상의 위치나 공간 정보를 다루기 위해서 만들어진 R-Tree(Guttman, a. 1984. R-Trees: A dynamic index structure for spatial searching)를 생물학 데이터에 적용한 결과이다. 공간 데이터를 가로만 있다고 하고 genome 정보를 검색하는데 사용하는 것이다.
간단하게 살펴보면, feature가 들어가는 일정한 폭의 bin(100Mb, 10Mb, 1Mb, 100Kb, 10Kb, 1Kb)에다가 float의 정수,소수 부분에 각각 폭(해당 feature가 속한 영역)과 번호(chromosome 상의 위치)를 넣게 된다. 그럼 1Kb 미만(bin=1000.012010)이고 영역이 1000(bin=10000.001201)인 feature를 검색한다면 검색의 범위가 좁아지기 때문에 훨씬 빠르게 원하는 위치의 원하는 만큼의 크기(길이)만을 가져올 수 있다(R-Tree 적용 후 그림 참고). R-tree는 gbrowse뿐만 아니라 UCSC Genome Browser에서도 사용하고 있다고 한다.
무턱대고 genome browser를 끄적 거리면서 고민했던 문제인데, 한번에 해결이 되어버리게 되었다. 좀 일찍 좀 찾아 볼 것을... 일전에 서열 유사성 검색에서 suffux tree와 함께 또하나의 Tree가 ^^;;
오늘은 요정도의 소개만 하고, 내일??은 실제 코드를 통해 어떻게 R-tree를 gbrowse에 적용해서 사용하는지에 대해서도 : )
반응형