티스토리 뷰
반응형
얼마전 자주 찾는 European Genetics and Anthropology Blog에 STRUCTURE를 이용한 분석 방법이 나와서 간단히 소개해 보려고 한다.
목적
자신의 유전정보가 과연 어느 민족과 유사한지 K-mean 클러스터링을 통해서 클러스터링한 결과를 알아보고자 한다. 향후 23andMe가 망하더라도 내 genotype 정보만으로 여러가지 분석을 지속적으로 수행할 수 있다.
준비물
1. 23andMe와 같은 유전자 검사 서비스 업체에서 자신의 유전자 검사 Raw 데이터가 필요하다. Raw 데이터는 다음과 같은 정보를 포함한다.
rs번호: 사람들이 가지고 있는 SNP에 대한 고유 ID (예: rs671 <-술먹으면 얼굴이 붉어짐에 관련한 SNP이라고 알려짐)
genotype: 해당 RS번호에 해당하는 자신의 genotype 정보(예:GG <-rs671이 GG타입인 경우 붉어지지 않는다고 알려짐)
2. 128개의 AIM SNP: 인간은 약 150만개 정도의 SNP을 가지고 있는데, 이중에서 128개의 SNP 만으로도 이 사람이 어느 민족의 사람인지를 구별할 수 있다고 한다. 이를 Ancestry Informative Marker 줄여서 AIM이라고한다.
3. 여러 민족들의 개개인의 128 AIM SNP에 대한 genotype 정보: 각 민족의 사람들의 128 AIM에 대한 정보
4. STRUCTURE 프로그램: K-mean 클러스터링을 이용해서 SNP genotype 데이터나 STR 데이터를 가지고 클러스터링을 수행하는 프로그램
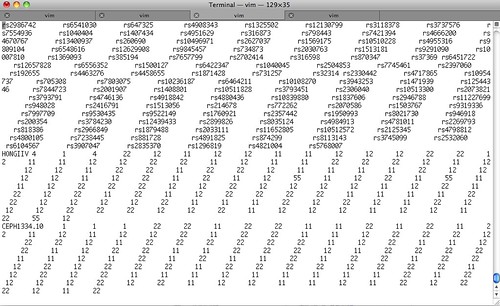
23andMe의 Raw 데이터를 STRUCTURE 포맷으로 변경해야 한다. 문자 형태의 genotype 정보는 숫자 형태로 변환되어야 하는데, 자신의 SNP 정보 중 rs731257이 AG 라면 12로 변경되어야 한다.(A=1, G=2) 128개의 AIM 중에 자신의 정보가 없는것이 있다면 해당 부분의 55로 바꾸어 준다. 일반적으로 -9-9를 쓰기도 합니다.
STRUCTURE 포맷으로 변환
STRUCTURE의 포맷은 첫행에 128개 SNP의 rs번호가 위치하고, 그 다음 행부터 각 개인의 rs번호에 해당하는 숫자화된 genotype 정보가 들어가게 된다. 그전에 총 4개의 컬럼이 더 필요한데, Label, PopID, Flag, Location 정보가 필요하다.
- Lable: 각 개인의 고유한 ID로 숫자 또는 문자 어떤것이든 상관없다.(예, CEPH1334.10)
- PopID: 개인이 속한 민족의 고유한 번호 (예, 중국인(CHB)인 경우 5, 유럽인(CEU)인 경우 1과 같이 자신이 직접 부여)
- Flag: 해당 PopID 정보를 STRUCTURE 프로그램 실행시 사용할 것인가?(1= 사용한다, 2= 사용하지 않는다.)
- Location: 해당 개인의 위치정보(예, 동아시아(EAS)인경우 1번, 유럽(EURA)인 경우 2번과 같이 자신이 직접 부여)
이렇게 각 행은 4개의 컬럼 + 128개의 SNP에 대한 genotype 정보를 숫자화한 값으로 구성되게 된다. 아래 그림은 3번에서 다운로드한 파일과 제 정보(HONGIIV 4 1 4 -> hongiiv라는 사람이 4번 민족(중국인)이며 1번(이정보를 프로그램에서 활용하며), 4번(동아시아)에 거주한다.)를 합친 STRUCTURE 포맷입니다.
STRUCTURE 실행하기
Step1
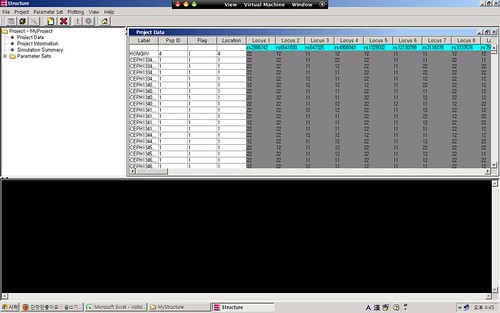
이제 STRUCTURE를 실행하고 새로운 프로젝트를 생성한다. 새 프로젝트를 생성하면 Project Wizard가 실행되며, 프로젝트의 이름, 프로젝트가 생성될 디렉토리, 전에 생성한 STRCUTRE 포맷의 파일을 각각 지정해 준다.
Step2
여기서 사용한 파일은 총 643명 + 1명(나) 정보이기 때문에 Number of individuals를 644, Ploidy of data: 1, Number of loci: 128, Missing data value: 55로 지정한다.
Step3
Row of marker names, Data file stores data for individuals in a single line을 각각 체크한다.
Step4
Individual ID for each individual, Putative population origin for each individual, USEPOPINFO selection flag, Sampling location information을 체크한다.
각 과정이 끝나면 다음과 같이 데이터가 로드된 것을 확인할 수 있다.
이제 실행하기 위한 파라메터를 지정하기 위해서 Parameter Sets> New를 선택한 후 Length of Burnin Period: 10000, Number of MCMC Reps after Burnin: 4000로 지정한다. 좀더 깔끔하고 정교한 결과를 위해 수치를 올려 줄 수도 있지만, 대신 시간이 많이 걸린다는 것 또한 명심해야 할것이다.
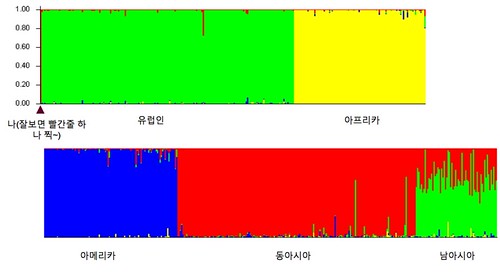
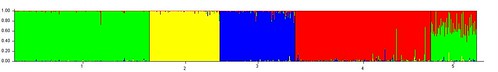
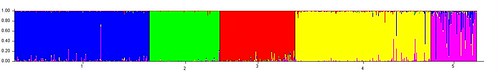
마지막으로 "!"나 Run 메뉴를 실행하면 K값을 물어보게 되는데, 적당히 4K-6K를 사용한다. 아래 결과는 K4(4개의 색상으로 각 개인을 구별)를 선택해서 실행한 결과이다. 나는 맨 처음 나오는 빨간줄로 동아시아인들(중국, 일본)과 같은 그룹에 속하는 것을 확인할 수 있다. 본 결과는 단지 128개의 SNP만을 사용한것으로 23andMe의 6십만개의 정보를 모두 활용한다면 좀더 세세한 결과를 볼 수 있을 것이다. 하지만 단지 128개 만으로도 인종을 구분할 수 있다는 것 자체도 흥미롭지 않을 수 없다.
첨부: 644명(나 포함)의 STRUCTURE 포맷 파일
첨부: K=4, K=5
목적
자신의 유전정보가 과연 어느 민족과 유사한지 K-mean 클러스터링을 통해서 클러스터링한 결과를 알아보고자 한다. 향후 23andMe가 망하더라도 내 genotype 정보만으로 여러가지 분석을 지속적으로 수행할 수 있다.
준비물
1. 23andMe와 같은 유전자 검사 서비스 업체에서 자신의 유전자 검사 Raw 데이터가 필요하다. Raw 데이터는 다음과 같은 정보를 포함한다.
rs번호: 사람들이 가지고 있는 SNP에 대한 고유 ID (예: rs671 <-술먹으면 얼굴이 붉어짐에 관련한 SNP이라고 알려짐)
genotype: 해당 RS번호에 해당하는 자신의 genotype 정보(예:GG <-rs671이 GG타입인 경우 붉어지지 않는다고 알려짐)
2. 128개의 AIM SNP: 인간은 약 150만개 정도의 SNP을 가지고 있는데, 이중에서 128개의 SNP 만으로도 이 사람이 어느 민족의 사람인지를 구별할 수 있다고 한다. 이를 Ancestry Informative Marker 줄여서 AIM이라고한다.
3. 여러 민족들의 개개인의 128 AIM SNP에 대한 genotype 정보: 각 민족의 사람들의 128 AIM에 대한 정보
4. STRUCTURE 프로그램: K-mean 클러스터링을 이용해서 SNP genotype 데이터나 STR 데이터를 가지고 클러스터링을 수행하는 프로그램
23andMe의 Raw 데이터를 STRUCTURE 포맷으로 변경해야 한다. 문자 형태의 genotype 정보는 숫자 형태로 변환되어야 하는데, 자신의 SNP 정보 중 rs731257이 AG 라면 12로 변경되어야 한다.(A=1, G=2) 128개의 AIM 중에 자신의 정보가 없는것이 있다면 해당 부분의 55로 바꾸어 준다. 일반적으로 -9-9를 쓰기도 합니다.
STRUCTURE 포맷으로 변환
STRUCTURE의 포맷은 첫행에 128개 SNP의 rs번호가 위치하고, 그 다음 행부터 각 개인의 rs번호에 해당하는 숫자화된 genotype 정보가 들어가게 된다. 그전에 총 4개의 컬럼이 더 필요한데, Label, PopID, Flag, Location 정보가 필요하다.
- Lable: 각 개인의 고유한 ID로 숫자 또는 문자 어떤것이든 상관없다.(예, CEPH1334.10)
- PopID: 개인이 속한 민족의 고유한 번호 (예, 중국인(CHB)인 경우 5, 유럽인(CEU)인 경우 1과 같이 자신이 직접 부여)
- Flag: 해당 PopID 정보를 STRUCTURE 프로그램 실행시 사용할 것인가?(1= 사용한다, 2= 사용하지 않는다.)
- Location: 해당 개인의 위치정보(예, 동아시아(EAS)인경우 1번, 유럽(EURA)인 경우 2번과 같이 자신이 직접 부여)
이렇게 각 행은 4개의 컬럼 + 128개의 SNP에 대한 genotype 정보를 숫자화한 값으로 구성되게 된다. 아래 그림은 3번에서 다운로드한 파일과 제 정보(HONGIIV 4 1 4 -> hongiiv라는 사람이 4번 민족(중국인)이며 1번(이정보를 프로그램에서 활용하며), 4번(동아시아)에 거주한다.)를 합친 STRUCTURE 포맷입니다.
STRUCTURE 실행하기
Step1
이제 STRUCTURE를 실행하고 새로운 프로젝트를 생성한다. 새 프로젝트를 생성하면 Project Wizard가 실행되며, 프로젝트의 이름, 프로젝트가 생성될 디렉토리, 전에 생성한 STRCUTRE 포맷의 파일을 각각 지정해 준다.
Step2
여기서 사용한 파일은 총 643명 + 1명(나) 정보이기 때문에 Number of individuals를 644, Ploidy of data: 1, Number of loci: 128, Missing data value: 55로 지정한다.
Step3
Row of marker names, Data file stores data for individuals in a single line을 각각 체크한다.
Step4
Individual ID for each individual, Putative population origin for each individual, USEPOPINFO selection flag, Sampling location information을 체크한다.
각 과정이 끝나면 다음과 같이 데이터가 로드된 것을 확인할 수 있다.
이제 실행하기 위한 파라메터를 지정하기 위해서 Parameter Sets> New를 선택한 후 Length of Burnin Period: 10000, Number of MCMC Reps after Burnin: 4000로 지정한다. 좀더 깔끔하고 정교한 결과를 위해 수치를 올려 줄 수도 있지만, 대신 시간이 많이 걸린다는 것 또한 명심해야 할것이다.
마지막으로 "!"나 Run 메뉴를 실행하면 K값을 물어보게 되는데, 적당히 4K-6K를 사용한다. 아래 결과는 K4(4개의 색상으로 각 개인을 구별)를 선택해서 실행한 결과이다. 나는 맨 처음 나오는 빨간줄로 동아시아인들(중국, 일본)과 같은 그룹에 속하는 것을 확인할 수 있다. 본 결과는 단지 128개의 SNP만을 사용한것으로 23andMe의 6십만개의 정보를 모두 활용한다면 좀더 세세한 결과를 볼 수 있을 것이다. 하지만 단지 128개 만으로도 인종을 구분할 수 있다는 것 자체도 흥미롭지 않을 수 없다.
첨부: 644명(나 포함)의 STRUCTURE 포맷 파일
첨부: K=4, K=5
K=4
반응형
공지사항
최근에 올라온 글