티스토리 뷰
반응형
HapMap GBrowse
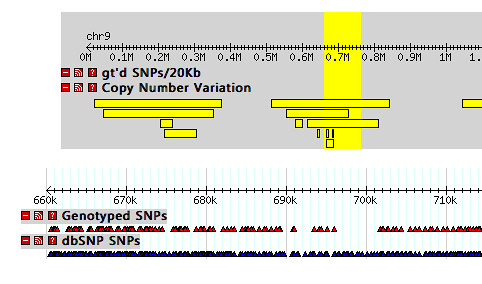
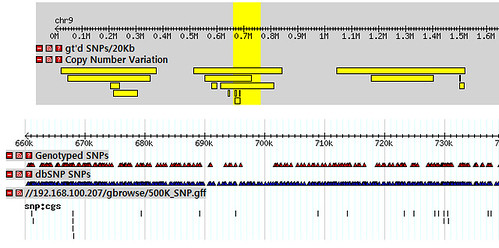
HapMap에서 사용하는 gbrowse에 내 데이터 살짝 얹어 보도록 하자. 직접 Genome Browser를 만들 수 없는 상황이라면 아주 유용하게 사용될 수 있을 것이다. 아래 그림은 chromosome 9의 0.7M 영역에서의 HapMap 프로젝트에서 Genotype한 SNP(붉은색 삼각형)들과 dbSNP의 SNP(파란색 삼각형)들을 GBrowse를 통해서 본 것이다.
여기서 내가 Genotype한 것들이 있을 경우 HapMap, dbSNP들과 비교해 보길 원한다면 자신의 Genotype한 데이터를 GFF 형식으로 만들어서 gbrowse에 살짝 넣어주면 된다. 자 그럼 시작해 보자.
GBrowse Hack
여기서는 다음의 3가지 방식을 통해서 GBrowse에 자신의 데이터를 표현하는 방법에 대해서 살펴보도록 하겠다.
우선 GFF3 포맷으로 SNP을 어떻게 표현할지 부터 알아보자. GFF는 다음과 같은 형식을 갖는 flat파일이다. 각각의 컬럼은 탭으로 구분되고 위의 형식에 따라 SNP를 표현하면 다음과 같다. GFF에 대한 상세한 Spec은 여기[1][2]를 참고 하면 된다.
seqid source type start end score strand phase attribute <-GFF3 형식
Chr1 dbsnp snp 200 200 . + . ID=SNP:rs1234;alleles:G/T <-GFF3 형식으로 SNP를 표현
Affymatirx에서 제공하는 500K SNP annotation 파일을 gff 형식으로 python 스크립트를 통해서 간단하게 변경한 후 gbrowse의 Add your own tracks에 gff 형식의 파일을 업로드한다.
HapMap에서 사용하는 gbrowse에 내 데이터 살짝 얹어 보도록 하자. 직접 Genome Browser를 만들 수 없는 상황이라면 아주 유용하게 사용될 수 있을 것이다. 아래 그림은 chromosome 9의 0.7M 영역에서의 HapMap 프로젝트에서 Genotype한 SNP(붉은색 삼각형)들과 dbSNP의 SNP(파란색 삼각형)들을 GBrowse를 통해서 본 것이다.
HapMap에서의 Genotyped SNP와 dbSNP SNPs 트랙
여기서 내가 Genotype한 것들이 있을 경우 HapMap, dbSNP들과 비교해 보길 원한다면 자신의 Genotype한 데이터를 GFF 형식으로 만들어서 gbrowse에 살짝 넣어주면 된다. 자 그럼 시작해 보자.
GBrowse Hack
여기서는 다음의 3가지 방식을 통해서 GBrowse에 자신의 데이터를 표현하는 방법에 대해서 살펴보도록 하겠다.
- gff 형식을 이용하는 방법
- gff 형식이 아닌 annotation 형식을 사용하는 방법
- 직접 DB에 넣은 후 gbrowse의 conf에서 어떻게 보여줄지 지정하는 방법
우선 GFF3 포맷으로 SNP을 어떻게 표현할지 부터 알아보자. GFF는 다음과 같은 형식을 갖는 flat파일이다. 각각의 컬럼은 탭으로 구분되고 위의 형식에 따라 SNP를 표현하면 다음과 같다. GFF에 대한 상세한 Spec은 여기[1][2]를 참고 하면 된다.
seqid source type start end score strand phase attribute <-GFF3 형식
Chr1 dbsnp snp 200 200 . + . ID=SNP:rs1234;alleles:G/T <-GFF3 형식으로 SNP를 표현
Affymatirx에서 제공하는 500K SNP annotation 파일을 gff 형식으로 python 스크립트를 통해서 간단하게 변경한 후 gbrowse의 Add your own tracks에 gff 형식의 파일을 업로드한다.
gff 파일 업로드하기
하단에 새롭게 생성된 500K SNP 트랙

하단에 새롭게 생성된 500K SNP 트랙
Anotation 사용하기
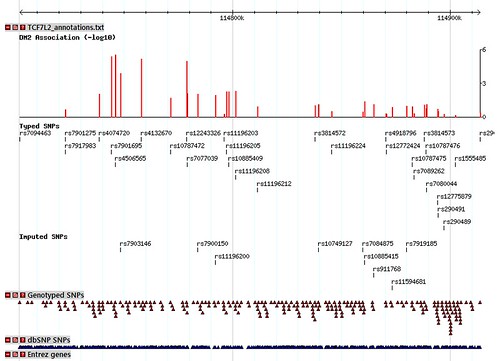
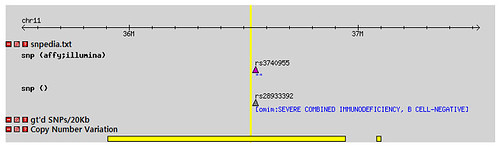
위의 gff 형식을 이용하면 해당 위치에 막대기( | )만 생겨서 별로 볼품이 없을 뿐더러 위치외에 어떤 정보를 표현해주지 않는다. 따라서 아래와 같은 annotation 파일을 만들면 어느정도 사용자의 취향 대로 정보를 표현할 수 있다. 아래 그림은 TCF7L2_annotations.txt와 snpedia의 두 annotation 파일을 위와 같이 upload your own data를 통해 업로드하여 HapMap 브라우저(GBrowse)상에서 확인한 모습이다. 좀 더 자세한 annotation 정보는 여기를 클릭
DB 사용하기
# nohup time ./script/load_gff --dsn dbi:mysql:hapmap_fin -gff_munge 500K_SNP.gff &
위의 명령어는 기존의 gbrowse 데이터베이스에 500K_SNP 정보를 새롭게 추가하는 load_gff 스크립트를 실행하는 명령어이다. gbrowse를 설치한 후 gff 파일들을 DB에 넣어주는 bulk_load_gff 명령어와 기존의 DB에 새로운 내용을 추가하는 load_gff 스크립트는 모두 Bio Perl 패키지에 들어있으며, HapMap 데이터의 경우에는 수정된 bulk_load_gff를 HapMap사이트에서 제공하고 있다. 참고로 기존의 HapMap 데이터가 들어있는 데이터베이스에 위의 명령어로 데이터를 추가하면 19시간이 걸렸다.(너무 오래 걸리네,,,)
HapMap 사이트에서 보면 bulk_load_gff 명령어로 gff 형식의 HapMap 데이터를 MySQL데이터베이스로 옮기는데 6~7시간, dbSNP 데이터를 로드하는데는 8~10시간이 걸린다고 한다.
bulk_load_gff : 6~7h(HapMap), 4h(테스트)
load_gff : 8~10h(HapMap), 7h(테스트)
위의 gff 형식을 이용하면 해당 위치에 막대기( | )만 생겨서 별로 볼품이 없을 뿐더러 위치외에 어떤 정보를 표현해주지 않는다. 따라서 아래와 같은 annotation 파일을 만들면 어느정도 사용자의 취향 대로 정보를 표현할 수 있다. 아래 그림은 TCF7L2_annotations.txt와 snpedia의 두 annotation 파일을 위와 같이 upload your own data를 통해 업로드하여 HapMap 브라우저(GBrowse)상에서 확인한 모습이다. 좀 더 자세한 annotation 정보는 여기를 클릭
Anotation 2
DB 사용하기
# nohup time ./script/load_gff --dsn dbi:mysql:hapmap_fin -gff_munge 500K_SNP.gff &
위의 명령어는 기존의 gbrowse 데이터베이스에 500K_SNP 정보를 새롭게 추가하는 load_gff 스크립트를 실행하는 명령어이다. gbrowse를 설치한 후 gff 파일들을 DB에 넣어주는 bulk_load_gff 명령어와 기존의 DB에 새로운 내용을 추가하는 load_gff 스크립트는 모두 Bio Perl 패키지에 들어있으며, HapMap 데이터의 경우에는 수정된 bulk_load_gff를 HapMap사이트에서 제공하고 있다. 참고로 기존의 HapMap 데이터가 들어있는 데이터베이스에 위의 명령어로 데이터를 추가하면 19시간이 걸렸다.(너무 오래 걸리네,,,)
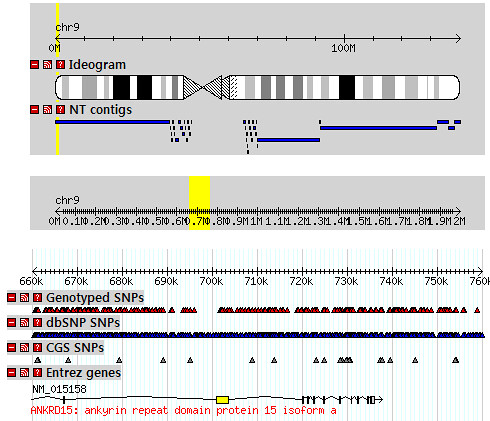
DB에 추가된 내용의 트랙(CGS SNPs)
HapMap 사이트에서 보면 bulk_load_gff 명령어로 gff 형식의 HapMap 데이터를 MySQL데이터베이스로 옮기는데 6~7시간, dbSNP 데이터를 로드하는데는 8~10시간이 걸린다고 한다.
bulk_load_gff : 6~7h(HapMap), 4h(테스트)
load_gff : 8~10h(HapMap), 7h(테스트)
반응형
공지사항
최근에 올라온 글