티스토리 뷰
반응형
UCSC, NCBI MapViewer, Ensembl과 같은 많은 Genome Browser들을 통해서 생물학 실험자들은 고급?의 프로그래밍 경험 없이도 genomic 영역을 시각화할 수 있지만 이들만을 가지고는 부족하기 때문에 여전히 프로그래밍과 데이터베이스를 다룰 줄 알아야 한다.
그러나 어느 정도까지는 프로그래밍 경험이 부족한 실험자들에게 서열데이터, 서열 정렬, 기능에 대한 주석등을 다룰 수 있도록 여러가지 방법을 통해 사용자들이 기존에 존재하는 소스로 부터 데이터를 모으고 간단한 인터페이스를 통해서 데이터를 결합, 계산, 원하는 것만 추출, 시각화, 서열 정렬, 결합, 삽입, 추출 등의 작업을 할 수 있게 해 줄 수 있도록 한다면 어떨까?
genome browser 및 기타 데이터 소스로 부터 데이터를 얻고, 간단한 조작을 통해서 자신이 원하는 작업을 할 수 있도록 해주는 large-scale genome 분석을 위한 플랫폼!! 바로 갤럭시가 전문적인 프로그래밍과 실험자를 연결해주는 역할을 하고 있다. Galaxy: A platform for interactive large-scale genome analysis
간단하게 UCSC의 테이블 브라우저를 통해 얻은 데이터에서 영역의 길이가 100이상인 것만 추출한다던가, start와 end값을 서로 뺀다던가 그러면 length가 나오겠죠 ^^ 뭐 이런 기초적인 작업에서 부터 다른 데이터 포맷으로의 변경, 간단한 통계 계산까지 자신의 작업을 하나하나 기록해가면서(History기능)이를 워크플로우화 할 수도 있다. 좀 나온지 오래된 논문이기는 하지만, 자신들이 연구하는 분야에 대한 데이터(데이터베이스) + 데이터시각화(브라우저쯤되겠죠) + 데이터조작(갤럭시 같은) = 하나의 플랫폼 정도 가지고 연구해야 하지 않을까??
그러나 어느 정도까지는 프로그래밍 경험이 부족한 실험자들에게 서열데이터, 서열 정렬, 기능에 대한 주석등을 다룰 수 있도록 여러가지 방법을 통해 사용자들이 기존에 존재하는 소스로 부터 데이터를 모으고 간단한 인터페이스를 통해서 데이터를 결합, 계산, 원하는 것만 추출, 시각화, 서열 정렬, 결합, 삽입, 추출 등의 작업을 할 수 있게 해 줄 수 있도록 한다면 어떨까?
genome browser 및 기타 데이터 소스로 부터 데이터를 얻고, 간단한 조작을 통해서 자신이 원하는 작업을 할 수 있도록 해주는 large-scale genome 분석을 위한 플랫폼!! 바로 갤럭시가 전문적인 프로그래밍과 실험자를 연결해주는 역할을 하고 있다. Galaxy: A platform for interactive large-scale genome analysis
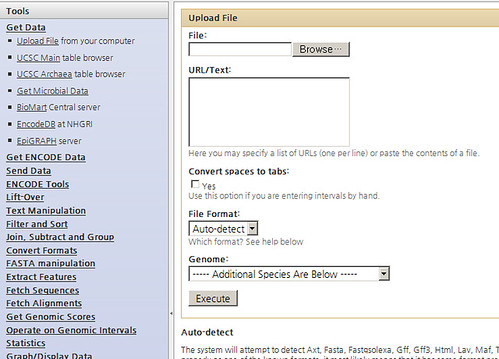
갤럭시에서의 데이터 가져오기 메뉴 - 사용자업로드 데이터, UCSC, BioMart, Encode 등 다양한 소스로 부터 데이터를 가져올 수 있다.
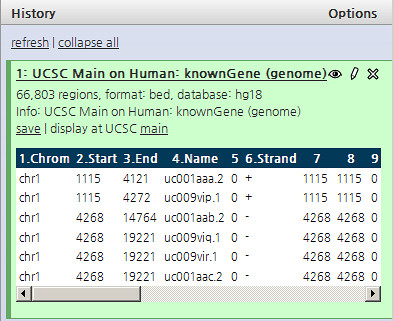
UCSC로 부터 known gene의 목록만을 가져온 화면


UCSC로 부터 known gene의 목록만을 가져온 화면

Filter 명령을 통해 위의 gene 목록중에서 chromosome 22번의 gene만을 추출하는 모습
간단하게 UCSC의 테이블 브라우저를 통해 얻은 데이터에서 영역의 길이가 100이상인 것만 추출한다던가, start와 end값을 서로 뺀다던가 그러면 length가 나오겠죠 ^^ 뭐 이런 기초적인 작업에서 부터 다른 데이터 포맷으로의 변경, 간단한 통계 계산까지 자신의 작업을 하나하나 기록해가면서(History기능)이를 워크플로우화 할 수도 있다. 좀 나온지 오래된 논문이기는 하지만, 자신들이 연구하는 분야에 대한 데이터(데이터베이스) + 데이터시각화(브라우저쯤되겠죠) + 데이터조작(갤럭시 같은) = 하나의 플랫폼 정도 가지고 연구해야 하지 않을까??
반응형
공지사항
최근에 올라온 글