티스토리 뷰
반응형
각 라면에 대하여 면발, 라면 그릇의 모양(크기), 국물맛에 대한 점수가 있다고 한다면, 3개의 변수(면, 그릇, 국물)를 가지고 PCA 분석을 해보자.
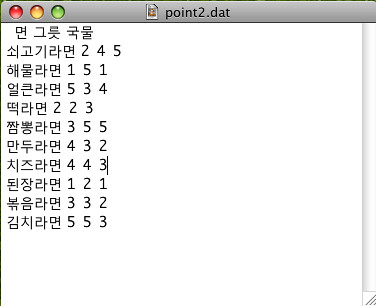
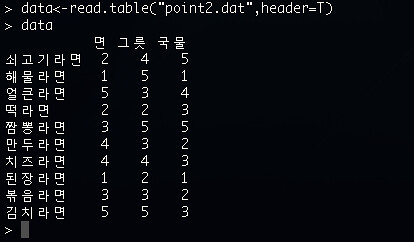
1. 데이터를 read.table()을 이용해서 로드한다.
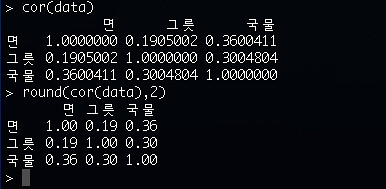
2. 이제 각 변수를 표준화하고, 이에 대한 상관행렬을 구한다.
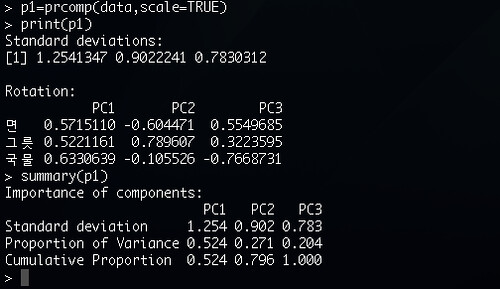
3. EigenValue, EigenVector 값을 구한다. 첫번째 주성분(PC1)의 기여율은 Cumulative Proportion이 0.524 즉, 52%의 기여율을 보인다. 이것은 PC1이 분석대상의 데이터가 가지고 있던 정보가 PC1 주성분에 어느정도 집약 되어 있는지에 대한 대략적인 크기가 된다.
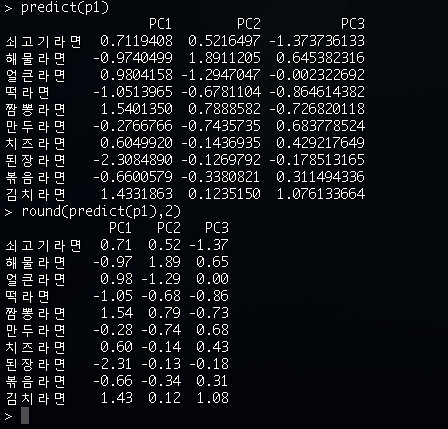
4. 각각에 대한 제1주성분, 제2주성분 점수를 구한다.
5. 제1주성분 점수와 제2주성분 점수를 토대로 그래프 작성
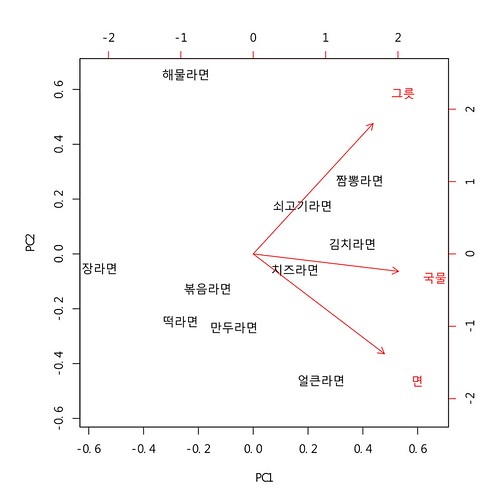
생성된 pdf 파일은 다음과 같다. 결론은 짬뽕, 김치, 쇠고기가 좋은 것으로 묶이고 국물이 역시 좋아야 한다는 결론,, -.-;;
1. 데이터를 read.table()을 이용해서 로드한다.
2. 이제 각 변수를 표준화하고, 이에 대한 상관행렬을 구한다.
3. EigenValue, EigenVector 값을 구한다. 첫번째 주성분(PC1)의 기여율은 Cumulative Proportion이 0.524 즉, 52%의 기여율을 보인다. 이것은 PC1이 분석대상의 데이터가 가지고 있던 정보가 PC1 주성분에 어느정도 집약 되어 있는지에 대한 대략적인 크기가 된다.
4. 각각에 대한 제1주성분, 제2주성분 점수를 구한다.
5. 제1주성분 점수와 제2주성분 점수를 토대로 그래프 작성
생성된 pdf 파일은 다음과 같다. 결론은 짬뽕, 김치, 쇠고기가 좋은 것으로 묶이고 국물이 역시 좋아야 한다는 결론,, -.-;;
반응형
공지사항
최근에 올라온 글