티스토리 뷰
반응형
본 글은 Alex Bisignano의 The Chromosome Chronicles 블로그의 "Identity by State SNP Analysis: Find Relatives, Test Paternity, and Determine Allele Sharing"이라는 글을 저자의 허락하에 한글화한 것으로 모든 권리는 Alex에게 있다. Thank you Alex : )

엘리사 로버슨과 조나단 펩스너의 지난 8월에 발행된 논문 "Visualization of Shared Genomic Regions and Meiotic Recombination in High-Density SNP Data" 에서 지놈상에서 두 사람이 공유하는 영역을 찾고 그 결과에 대한 내용이 실려있다.
가족 관계에서 얼마나 많은 게놈 유사성을 가지고 있는지에 대한 것은 지금까지 많은 방법이 존재한다(IBD, identity by decent 방법) 로버슨과 펩스너는 SNP를 기반으로 하는 identity by state(IBS)를 통해서 어떠한 가족 정보도 가지지 않는 두 사람을 비교하는 방법에 대해서 이야기하고 있다.
Identity by State(IBS)를 이용한 게놈 비교: 개요
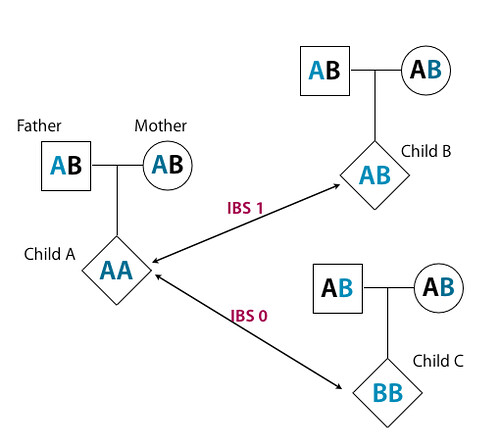
IBS는 두 사람의 SNP을 비교해서 다음의 세 분류로 나누게 된다.
1. Identical: 두 사람은 같은 지노타입을 가지고 있다.(예, AA와 AA, BB와 BB, AB와 AB처럼)
2. One-Allele Shared: 두 사람간에 하나만 같은 것을 공유한다. (예, AA와 AB, AB와 BB처럼)
3. No alleles shared: 둘다 같지 않다(예, AA와 BB 처럼)
SNP을 가지고 IBS를 통해서 얻을 수 있는 정보는 이것 밖에 얻을것이 없다. 이러한 정보는 유전체 전장을 커버하는 고밀도 SNP을 통해서 얻을 수 있다(이미 23andMe 사용자들은 이러한 정보를 가지고 있으며, Illumina나 Affymetrix 플랫폼들이 유전체 전장을 커버한다).
이 논문에서는 IBS의 값이 identical인 경우 2, one-allele shared인 경우 1, no allels shared인 경우 0으로 표시하고 있다. 각 염색체별로 각기 다른 3개의 상태를 ideogram 기반으로 IBS 값을 표시하고 있다. 다음은 논문의 그림을 보여주고 있다.
보는 바와 같이, 염색체 10번에 대한 그림으로 두 명의 부모(아버지-어머니, 두 사람은 0촌으로 아무런 unrelated)에 대한것으로 2, 1, 0 순서로 많음을 보여주고 있다. 2와 1, 0이 혼재해 있다는 것은 두 사람은 친인척 관계가 적다라고 할 수 있다. 또한 2번 화살표와 같이 0이 존재하지 않는 영역은 유심히 살펴 봐야할 필요가 있다.
IBS 값 0이 존재하지 않는다는 것(공유하지 않는 SNP이 없다는 것)은 두 사람이 actual haplotype을 공유한다는 증거이다. 이 예에서는 0값이 존재하지 않는 영역에 1이나 2의 값은 존재한다. 이것은 두 사람이 이 지역에 대해서 하나의 haplotype을 공유한다는것을 가르킨다. 다음 표는 어느 한 지역에서 IBS 값을 어떤 의미로 받아 들여야 하는지를 보여준다.
로버슨과 펩스너는 계속해서 인척 관계에서 IBS를 보여주고 있다.
어머니와 아들의 비교 결과를 보면 IBS 값이 2와 1만 보이는 것을 확인할 수 있다. 당연히 아들은 어머니와 아버지에게서 각각 1개씩 물려받기 때문에 적어도 1개이상은 같은 즉 1 이상의 IBS 값을 가져야만 한다. 그렇지 않고 0이 나온 경우 이것은 지노타이핑 에러 이거나 rare 또는 돌연변이 일 수 있다.
두번째 형제의 비교는 좀더 복잡한 양상을 보이지만 좀 더 많은 정보를 내포하고 있다. 위 형제의 비교에서는 2, 1 , 0의 모든 값이 나타나는데 이것은 자식들이 부모로부터 각각 서로 다른 allele를 받았다는 것을 의미하는 것으로 재조합이 일어났다는 것을 의미한다. 이것이 바로 같은 형제이지만 서로 다른 이유가 된다. 멋지지 않은가!
IBS 분석을 통해서 우리는 무엇을 알 수 있는가
우리는 IBS 분석을 통해서 재조합과 형제간 allele 공유를 보았다. 이것은 매우 유용한 정보로 IBS가 유용하게 쓰이는 다른 몇가지 사례를 보도록 하겠다.
GenomicRelator: IBS 분석 구현
새벽 3시 이 프로그램을 만들었으며, 이것은 free로 한다. 왜냐면 난 쿨하기 때문이다. 또한 이 프로그램을 상용으로 사용하고자 할 경우 필자에게 반드시 연락해주기 바란다.
Genome Relator 프로그램은 다음의 두가지 기능을 가지고 있다.
프로그램의 실행
본 프로그램은 여기서 다운로드가 가능하며, 이것은 압축된 ZIP 파일 형태로 4개의 JAR 파일로 구성되어 있다. 이 프로그램을 사용하기 위해서는 압축을 해제한 후 비교하고자 하는 게놈 파일(file1.txt, file2.txt로 저장된)과 같은 폴더에 저장한다. 그런 후 JAR 파일을 더블 클릭하여 실행한다. 프로그램이 실행되기 위해서는 Java Runtime Environment가 설치되어 있어야 하며, 충분한 메모리가 준비되어야 한다.
GenomeRelatorRAW 는 두개의 파일을 비교하여 IBS 값을 다음과 같은 그림으로 보여준다.

그림만으로 별다른 해석없이 이해가 가능하며, 23andMe 파일의 경우 20분 정도의 시간이 소요된다.
일반적인 버전의 GenomeRelator는 매끄럽게 표현이 가능하며, 다음과 같은 그림을 얻을 수 있다.

이것은 한번에 250개의 SNPs의 영역에서 1%이상의 "0"과 5%이상의 "1"값이 존재한다면 그 지역은 2,1,0(위 표 참고) 즉 관계가 없음을 의미하며, 1%이하의 "0"과 5%이상의 "2"와 "1"값을 가지면 2,1(하나의 Allele를 공유), 1%이하의 "0"과 5%이상의 "2"가 있다면 2(두개의 Allele)를 공유한다고 가정하고 그림을 그리게 된다. 따라서 아래의 그림처럼 좀 더 단순한, 매끄러운 그림을 보여주게 된다. 250, 1%, 5%의 값은 향후 GUI 버전에서 변경할 수 있도록 한다.
입력 파일 생성 규칙
프로그램 실행 결과
일반적인 버전을 실행하면 단지 그림뿐만 아니라 몇개의 파일도 같이 생성된다. 각 염색체별 텍스트 파일이 같이 생성되며, 이것은 분석을 하기 위한 중간 단계에서 생성되는 파일이다. "IBS Summary"라는 각 염색체별 파일 또한 생성되는데, 이 파일에는 영역( 한번에 250 SNPs) 의 시작과 끝을 보여준다.
프로그램의 공개
본 프로그램은 반드시 www.chromosomechronicles.com을 통해서만 배포되어야 한다. 또한 교육이나 재미를 위해서는 공짜이며, 어떠한 피드백이나 에러 리포트에 대해서 감사하게 생각한다.

가족 관계에서 얼마나 많은 게놈 유사성을 가지고 있는지에 대한 것은 지금까지 많은 방법이 존재한다(IBD, identity by decent 방법) 로버슨과 펩스너는 SNP를 기반으로 하는 identity by state(IBS)를 통해서 어떠한 가족 정보도 가지지 않는 두 사람을 비교하는 방법에 대해서 이야기하고 있다.
Identity by State(IBS)를 이용한 게놈 비교: 개요
IBS는 두 사람의 SNP을 비교해서 다음의 세 분류로 나누게 된다.
1. Identical: 두 사람은 같은 지노타입을 가지고 있다.(예, AA와 AA, BB와 BB, AB와 AB처럼)
2. One-Allele Shared: 두 사람간에 하나만 같은 것을 공유한다. (예, AA와 AB, AB와 BB처럼)
3. No alleles shared: 둘다 같지 않다(예, AA와 BB 처럼)
SNP을 가지고 IBS를 통해서 얻을 수 있는 정보는 이것 밖에 얻을것이 없다. 이러한 정보는 유전체 전장을 커버하는 고밀도 SNP을 통해서 얻을 수 있다(이미 23andMe 사용자들은 이러한 정보를 가지고 있으며, Illumina나 Affymetrix 플랫폼들이 유전체 전장을 커버한다).
이 논문에서는 IBS의 값이 identical인 경우 2, one-allele shared인 경우 1, no allels shared인 경우 0으로 표시하고 있다. 각 염색체별로 각기 다른 3개의 상태를 ideogram 기반으로 IBS 값을 표시하고 있다. 다음은 논문의 그림을 보여주고 있다.
보는 바와 같이, 염색체 10번에 대한 그림으로 두 명의 부모(아버지-어머니, 두 사람은 0촌으로 아무런 unrelated)에 대한것으로 2, 1, 0 순서로 많음을 보여주고 있다. 2와 1, 0이 혼재해 있다는 것은 두 사람은 친인척 관계가 적다라고 할 수 있다. 또한 2번 화살표와 같이 0이 존재하지 않는 영역은 유심히 살펴 봐야할 필요가 있다.
IBS 값 0이 존재하지 않는다는 것(공유하지 않는 SNP이 없다는 것)은 두 사람이 actual haplotype을 공유한다는 증거이다. 이 예에서는 0값이 존재하지 않는 영역에 1이나 2의 값은 존재한다. 이것은 두 사람이 이 지역에 대해서 하나의 haplotype을 공유한다는것을 가르킨다. 다음 표는 어느 한 지역에서 IBS 값을 어떤 의미로 받아 들여야 하는지를 보여준다.
| IBS 영역 |
의미 |
| 2, 1, 0이 모두 존재하는 지역 |
아무런 Allele를 공유하고 있지 않음 |
| 2, 1이 존재하는 지역 |
하나의 Allele를 공유 |
| 2만 존재하는 지역 |
두 개의 Allele를 공유 |
로버슨과 펩스너는 계속해서 인척 관계에서 IBS를 보여주고 있다.
어머니와 아들의 비교 결과를 보면 IBS 값이 2와 1만 보이는 것을 확인할 수 있다. 당연히 아들은 어머니와 아버지에게서 각각 1개씩 물려받기 때문에 적어도 1개이상은 같은 즉 1 이상의 IBS 값을 가져야만 한다. 그렇지 않고 0이 나온 경우 이것은 지노타이핑 에러 이거나 rare 또는 돌연변이 일 수 있다.
두번째 형제의 비교는 좀더 복잡한 양상을 보이지만 좀 더 많은 정보를 내포하고 있다. 위 형제의 비교에서는 2, 1 , 0의 모든 값이 나타나는데 이것은 자식들이 부모로부터 각각 서로 다른 allele를 받았다는 것을 의미하는 것으로 재조합이 일어났다는 것을 의미한다. 이것이 바로 같은 형제이지만 서로 다른 이유가 된다. 멋지지 않은가!
IBS 분석을 통해서 우리는 무엇을 알 수 있는가
우리는 IBS 분석을 통해서 재조합과 형제간 allele 공유를 보았다. 이것은 매우 유용한 정보로 IBS가 유용하게 쓰이는 다른 몇가지 사례를 보도록 하겠다.
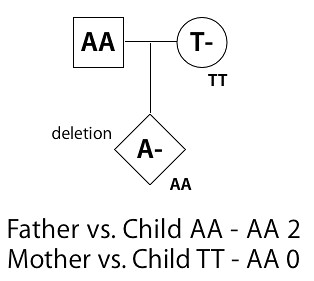
- Hemizygous Deletion 검사: 앞서서 부모와 자식간에는 0이 나올 수 없다는 것을 보았는데, 만약 0이 나온다면 이것은 아래 그림고 같이 deletion이 일어났다는 것을 의미한다. 아버지의 경우 "AA", 어머니의 경우 하나의 "T" 하나만 가지고 다른 Allele가 없는 경우(genotype은 "TT"로 나옴) 이렇게 된 경우 자식은 "A" Allele와 deletion된 것을 각각 물려 받아서 "A-"이지만 genotype은 AA가 나오기 때문에 부모 중 어머니와 비교 할 경우 적어도 1이 나와야 하지만, "TT"와 "AA" 이기 때문에 0이 나오게 되며, 이를 Hemizygous Deletion이라고 한다.
- 친인척의 구분: IBS는 친인척 관계를 규명하는 척도로 사용될 수 있는데, 친인척 관계가 있는 경우 좀 더 많은 Allele를 공유하기 때문이다.
Deletion

다음의 프로그램은 염색체상에 색상을 통해서 Allele의 공유정도를 보여주는데, 회색 영역은 SNP 정보가 없는 경우, 검정색인 경우 공유하는 Allele가 없는 경우, 빨간색은 하나의 Allele를 공유하는 경우, 초록색은 두개의 Allele를 공유하는 것을 의미한다. 다음의 그림은 혈연관계는 없지만, 공통의 선조를 가진 즉 같은 인종의 사람임을 보여준다.
GenomicRelator: IBS 분석 구현
새벽 3시 이 프로그램을 만들었으며, 이것은 free로 한다. 왜냐면 난 쿨하기 때문이다. 또한 이 프로그램을 상용으로 사용하고자 할 경우 필자에게 반드시 연락해주기 바란다.
Genome Relator 프로그램은 다음의 두가지 기능을 가지고 있다.
- 일반적인 IBS 계산: 본 프로그램은 두개의 게놈 파일을 읽고 각 염색체에 대한 IBS 값을 계산 한후 그림을 그려준다.(녹색/빨강/검정)
- IBS 데이터의 매끄러운 표현: 각 영역이 가지고 있느너 0, 1, 2에 대한 값을 가지고 쉽게 이해할 수 있도록 그림으로 제공한다.
프로그램의 실행
본 프로그램은 여기서 다운로드가 가능하며, 이것은 압축된 ZIP 파일 형태로 4개의 JAR 파일로 구성되어 있다. 이 프로그램을 사용하기 위해서는 압축을 해제한 후 비교하고자 하는 게놈 파일(file1.txt, file2.txt로 저장된)과 같은 폴더에 저장한다. 그런 후 JAR 파일을 더블 클릭하여 실행한다. 프로그램이 실행되기 위해서는 Java Runtime Environment가 설치되어 있어야 하며, 충분한 메모리가 준비되어야 한다.
GenomeRelatorRAW 는 두개의 파일을 비교하여 IBS 값을 다음과 같은 그림으로 보여준다.

그림만으로 별다른 해석없이 이해가 가능하며, 23andMe 파일의 경우 20분 정도의 시간이 소요된다.
일반적인 버전의 GenomeRelator는 매끄럽게 표현이 가능하며, 다음과 같은 그림을 얻을 수 있다.

이것은 한번에 250개의 SNPs의 영역에서 1%이상의 "0"과 5%이상의 "1"값이 존재한다면 그 지역은 2,1,0(위 표 참고) 즉 관계가 없음을 의미하며, 1%이하의 "0"과 5%이상의 "2"와 "1"값을 가지면 2,1(하나의 Allele를 공유), 1%이하의 "0"과 5%이상의 "2"가 있다면 2(두개의 Allele)를 공유한다고 가정하고 그림을 그리게 된다. 따라서 아래의 그림처럼 좀 더 단순한, 매끄러운 그림을 보여주게 된다. 250, 1%, 5%의 값은 향후 GUI 버전에서 변경할 수 있도록 한다.
입력 파일 생성 규칙
- 입력 파일은 반드시 file1.txt와 file2.txt.로 지정한다. 그리고 반드시 프로그램과 같은 폴더(jar 파일이 존재하는)에 있어야 한다.
- 입력 파일은 한 줄의 헤더 부분을 가지고 있어야 한다.
- 각각 두개의 입력 파일은 동일한 SNP을 가지고 있어야 한다.
- 입력 파일은 rsid, chromosome, position, genotype의 4개의 컬럼을 가지고 있어야 한다. genotype은 하나 또는 2개의 문자로 구성한다.
프로그램 실행 결과
일반적인 버전을 실행하면 단지 그림뿐만 아니라 몇개의 파일도 같이 생성된다. 각 염색체별 텍스트 파일이 같이 생성되며, 이것은 분석을 하기 위한 중간 단계에서 생성되는 파일이다. "IBS Summary"라는 각 염색체별 파일 또한 생성되는데, 이 파일에는 영역( 한번에 250 SNPs) 의 시작과 끝을 보여준다.
프로그램의 공개
본 프로그램은 반드시 www.chromosomechronicles.com을 통해서만 배포되어야 한다. 또한 교육이나 재미를 위해서는 공짜이며, 어떠한 피드백이나 에러 리포트에 대해서 감사하게 생각한다.
반응형
공지사항
최근에 올라온 글